Crean un modelo computacional que simula el crecimiento biomecánico de los tumores de mama
Una investigación de la UC3M y la Johns Hopkins University
20/03/24
Científicos de la Universidad Carlos III de Madrid (UC3M) y de la Johns Hopkins University (JHU), en EEUU, han analizado el crecimiento de los tumores de mama desde una perspectiva biomecánica y han creado un modelo computacional que simula el proceso de invasión de las células cancerosas, en función de las características del tejido circundante y de las uniones celulares, entre otros parámetros. Este tipo de modelos permitirán ayudar a predecir el progreso de un tumor en pacientes a partir de las propiedades mecánicas (la rigidez, densidad, etc) del área donde se desarrolla, que se pueden conocer a través de una biopsia o técnicas de imagen.
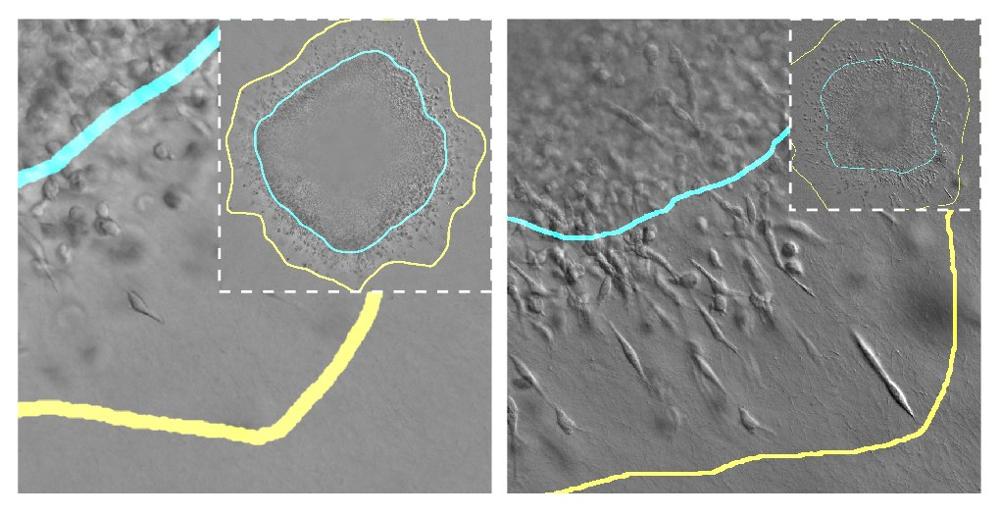
Células tumorales invadiendo el tejido circundante bajo distintas condiciones mecánicas. La línea azul indica el contorno del tumor sólido y la amarilla el espacio invadido por células tumorales que se desprenden del tumor.
El proceso de crecimiento de un tumor sólido implica su expansión a través del tejido circundante, compuesto habitualmente de una matriz fibrilar (por ejemplo, colágeno). Su expansión depende de muchos factores, como el número total de células del tumor, su volumen y rigidez, su acceso a nutrientes y las propiedades mecánicas del tejido donde se desarrolla. Apoyados en modelos experimentales in vitro, estos investigadores de la UC3M y de la JHU han desarrollado un modelo que permite simular en un ordenador el crecimiento de tumores teniendo en cuenta estos factores. “En este modelo hemos simulado cómo las células de un tumor de mama se multiplican e invaden el tejido a su alrededor, y cómo se reproducen más o menos según cómo de rígido y poroso sea el tejido alrededor o cómo de fuertes sean las uniones de unas células con otras”, explica uno de los investigadores, Daniel García González, Profesor Titular del Dpto. de Medios Continuos y Teoría de Estructuras de la UC3M y responsable del proyecto ERC 4D-BIOMAP.
Para ello, los investigadores han trabajado con esferoides, que son agrupaciones de células tumorales con diferentes características que están embebidas en una matriz y que funcionan como un modelo, simulando cómo se comportan las células en un tumor real. “Son sistemas muy potentes que se están utilizando cada vez más para hacer estudios sobre el comportamiento de los tumores y también para hacer estudios de posibles terapias”, explica otra de las investigadoras, Arrate Muñoz-Barrutia, catedrática en el Departamento de Bioingeniería de la UC3M.
Gracias a estos esferoides, los investigadores han podido modificar en el laboratorio ciertos aspectos biológicos o mecánicos de estos tumores y evaluar cómo estas variables influyen en la proliferación y migración de las células. Posteriormente, han transformado estas observaciones en ecuaciones matemáticas que han implementado en un modelo computacional. De esta manera, podían comprobar en paralelo (en el simulador en el ordenador y en el modelo experimental con los esferoides en el laboratorio) el comportamiento de las variables que afectan al crecimiento de estos tumores. “Nuestros nuevos esferoides con varios compartimentos nos permitieron controlar y ajustar las propiedades biomecánicas del sistema controlando la densidad de colágeno y la expresión de E-cadherina, que se sabe que desempeñan un papel en la progresión del cáncer de mama. Fue muy emocionante trabajar con este equipo y ver el desarrollo de estos procesos desde perspectivas tanto experimentales como computacionales”, indica otro de los autores del estudio, Denis Wirtz, del Departamento de Ingeniería Química y Biomolecular de la JHU.
“Mientras que experimentalmente la proliferación y la invasión a menudo se miden como dos parámetros independientes, observamos un fuerte acoplamiento de estos procesos. Aunque el análisis de estas contribuciones no se podían desacoplar utilizando aproximaciones experimentales tradicionales, el modelo computacional nos permitió estudiar estos procesos de manera independiente y obtener información sobre las propiedades biomecánicas de nuestro sistema”, añade otra de las investigadoras del equipo de la JHU, Ashleigh Crawford.
Las aplicaciones futuras de este estudio resultan prometedoras, según los investigadores. “Si sabemos cuáles son los parámetros mecánicos que afectan a que el tumor crezca más o menos, entonces podríamos utilizar esos datos para mejorar el tratamiento o desarrollar nuevos fármacos a medio o largo plazo”, comenta Daniel García González. “Pensamos que estos estudios abren las puertas a desarrollos de tecnologías que permitan caracterizar la mecánica del tumor, lo que puede añadir información relevante para la elección de terapia para el cáncer”, añade Arrate Muñoz-Barrutia.
El equipo de científicos también resalta la importancia de la investigación multidisciplinar en este caso, puesto que se han realizado aportaciones tanto desde el ámbito computacional y matemático hasta el puramente biológico. “Mi formación como ingeniera biomédica, estudiando en la UC3M, me ha permitido poder colaborar en todas las partes de esta investigación y crear puentes de comunicación entre disciplinas, que emplean terminologías distintas”, apunta otra de las autoras del estudio, Clara Gómez Cruz, doctoranda del Dpto. de Medios Continuos y Teoría de Estructuras y de la UC3M.
Esta investigación se enmarca dentro de 4D-BIOMAP (Biomechanical Stimulation based on 4D Printed Magneto-Active Polymer; Estimulación Biomecánica basada en Polímeros Magneto-Activos por impresión 4D), un proyecto financiado por el Consejo Europeo de Investigación (European Research Council) a través de una ayuda ERC Starting Grant del Programa Marco de Investigación e Innovación de la Unión Europea, Horizonte 2020 (GA 947723). Además, ha recibido financiación por parte del Instituto Nacional de Salud (National Institute of Health) y el Instituto Nacional del Cáncer (National Cancer Institute) de EEUU.
Referencia bibliográfica:
Crawford A.J. Gomez-Cruz, C. Russo G. C. Huang, W. Bhorkar I. Roy, T, Muñoz-Barrutia, A. Wirtz, D. Garcia-Gonzalez, D. (2024). Tumor proliferation and invasion are intrinsically coupled and unraveled through tunable spheroid and physics-based models. Acta Biomaterialia, Volume 175, Pages 170-185, ISSN 1742-7061. https://doi.org/10.1016/j.actbio.2023.12.043

